GTEx数据库是一个广泛收集了来自449名健康人类捐赠者的7000多份尸检样本的数据集,涵盖了44种不同组织类型,包括31个实体器官组织10个脑分区全血以及来自捐赠者血液和皮肤的细胞系利用这些数据,可以深入研究基因表达在不同组织和个体之间的差异数据准备阶段,首先,详解人类基因在不同数据库中的ID我们可以通过GTEx官网或UCSC xene详解人类基因在不同数据库中的ID;注释工具和方法包括1 clusterProfiler包中的bitr函数适用于基因芯片分析和GOKEGGGSEA2 biomaRt工具通过连接到Ensembl的人类数据库,获取基因和转录本的对应关系3 GTF文件通过加载预先处理过的GTF数兆陪据,直接与ENST ID匹配,获取详细的基因注释在进行基因注释时,请确保数据的;代表该序列在UniRef数据库中的编号,HBA_HUMAN表示简称,Hemoglobin subunit。
医学大数据及其综合分析四“大数据”在医学领域的重要性日益凸显,而GTEx数据库作为一类优质的人类数据集,其应用在科研转化与成果验证中显得尤为重要通过GTEx数据库,我们可以探索人类基因表达的相关性,尤其是将动物模型研究发现的蛋白相互作用验证于人类肝脏上在科学研究中,动物或细胞模型常被用以;输入差异基因列表,确保只有蓝色打钩的基因可用去除红色和空白圈圈的基因,保留150个基因点击submit04 等待结果,通常需要半小时以上查看项目下载文件05 使用HEATMAP查看热图形式的数据显示,列代表不同的cell ID,行代表Perturbagen,注释包含数据库来源名称描述不同score阈值条件下的占比以及。
首先,登录DAVID官网,你会在左侧功能菜单栏看到四个主要工具功能注释功能分类基因ID转换和基因名称查看功能注释是常用工具,用于进行分析进入后,上传你的差异基因列表,每个基因ID以逗号分隔,注意列表不超过3000个基因选择基因类型后,可能需要指定物种以人类为例,选择homo sapiens,并选择。
生信技能树R专辑11实践与感想的主要内容包括以下几点研究内容利用R语言获取人类基因信息该专辑探讨了如何使用R语言来获取人类基因的详细信息,这些信息包括基因的名称ID符号以及别名比较不同数据库中的基因编码方式通过研究,专辑详细分析了NCBI的Entrez gene IDHUGO symbolRefSeq ID以及;3 **尝试babelgene包**针对R语言用户,babelgene包提供了一种直接便捷的解决方案,用于基因ID的转换,特别适合于处理复杂的人类基因组数据,且不受联网不稳定和旧版本库更新限制的影响结语 基因ID转换是转录组分析中的重要步骤,通过本文分享的方法和工具,研究者能够更高效地在不同物种间进行基因;直接下载GEO数据集时,获取表达矩阵的步骤可能会遇到瓶颈虽然下载的Series Matrix Files中通常只有探针编号,缺少对应的基因名称等信息,但接下来的步骤能解决这一问题访问GEO官网的Platforms部分,了解不同测序集所采用的人类基因组版本在本例中,数据集使用了Human Genome U133 Set的AB版本获取;本文探讨利用R语言获取人类基因的详细信息,包括名称ID符号以及别名通过研究,我们了解了不同数据库中基因编码方式的异同首先,我们关注的是NCBI的Entrez gene ID此ID在NCBI的Entrez基因数据库中使用,用于标识不同物种的基因值得注意的是,同一基因在不同物种下的Entrez ID不同,但这一ID相对。
首先,我们可以使用NCBI的homologene工具,访问网址选择需要查找的基因例如TP53,输入基因名字后搜索,表格将显示该基因在不同物种中的同源基因名称另一种方法是使用R包homologene,它特别适合处理大量基因的同源查找问题通过R代码批量处理,可以高效地获取成百上千;数据库对象与标识符KEGG ID是KEGG数据库中用于区分不同数据库对象的独特标识符例如,基因使用K numbers进行标识,而化合物则使用KEGG COMPOUND进行标识功能与应用KEGG数据库帮助科研人员更好地理解和分析基因功能特别是对于生物信息学研究者来说,理解KEGG通路图至关重要,它提供了关于代谢和调控;基因集富集分析GSEA是一种研究方法,旨在确定预先定义的基因集是否在不同生物状态之间显示出显著一致的差异它需要基因列表与功能基因集,常用的数据集来源包括MSigDB中的预定义功能基因集数据集包含两种ID版本,即SYMBOL ID与ENtrez ID版本具体而言,KEGG基因集存储在C2目录下,而GO基因集位于C5目录;1 bioDBnet的db2db工具生物数据库网络提供了一个集成大量生物数据库的应用程序,包括GeneUniProtEnsemblGOAffyRefSeq等其db2db工具能帮助在不同数据库间进行ID转换,转换结果可导出为Excel表格或txt文件2 Uniprot的ID mapping工具Uniprot数据库ID与其他数据库ID的相互转换可以使用此工具;对于非编程用户,网站工具如biodbnet可以便捷地进行转换输入人类基因symbol,获取对应ID,但这个方法速度慢,且并非所有symbol都能找到对应ID,因此对于会编程的用户来说,更推荐使用编程方法编程转换中,R语言的模块是常见选择安装后,通过SQLite文件存储的数据,可以查看包含多个数据表,如;Ensembl数据库的基因命名,如ENSG00000,由物种类型和特定编号组成,有时版本号会被省略人类基因ID通常省略物种和版本,如ENSG00000转换工具BioMart与bioDBnet 在基因ID的转换和查询中,我们有强大的工具支持例如,Ensemble的Web工具BioMart,不仅提供了基因功能注释,还支持跨数据库的ID转换。
NC表示人类基因组DNA的RefSeq链接序列NM表示mRNA的RefSeqNP表示蛋白质的RefSeq 查找基因的基本信息的方法如下根据文献中已知的基因ID如果你在文献中看到你感兴趣的基因,而且文中还提到了 基因在Genbank中的ID号,那就好办了,直接打开 ,在Search后的下拉框中选择;SNP的诞生与运作 DNA,生物遗传信息的载体,携带着30亿对碱基对,其中约5000万个SNP,赋予了我们独特的遗传指纹SNP是DNA序列中最常见的变异形式,它们是基因组中等位基因的舞台,一个位点上拥有两个不同的核苷酸,一个为主流,另一个为罕见,共同塑造了我们的遗传特性SNP的连锁不平衡,即等位基因在。
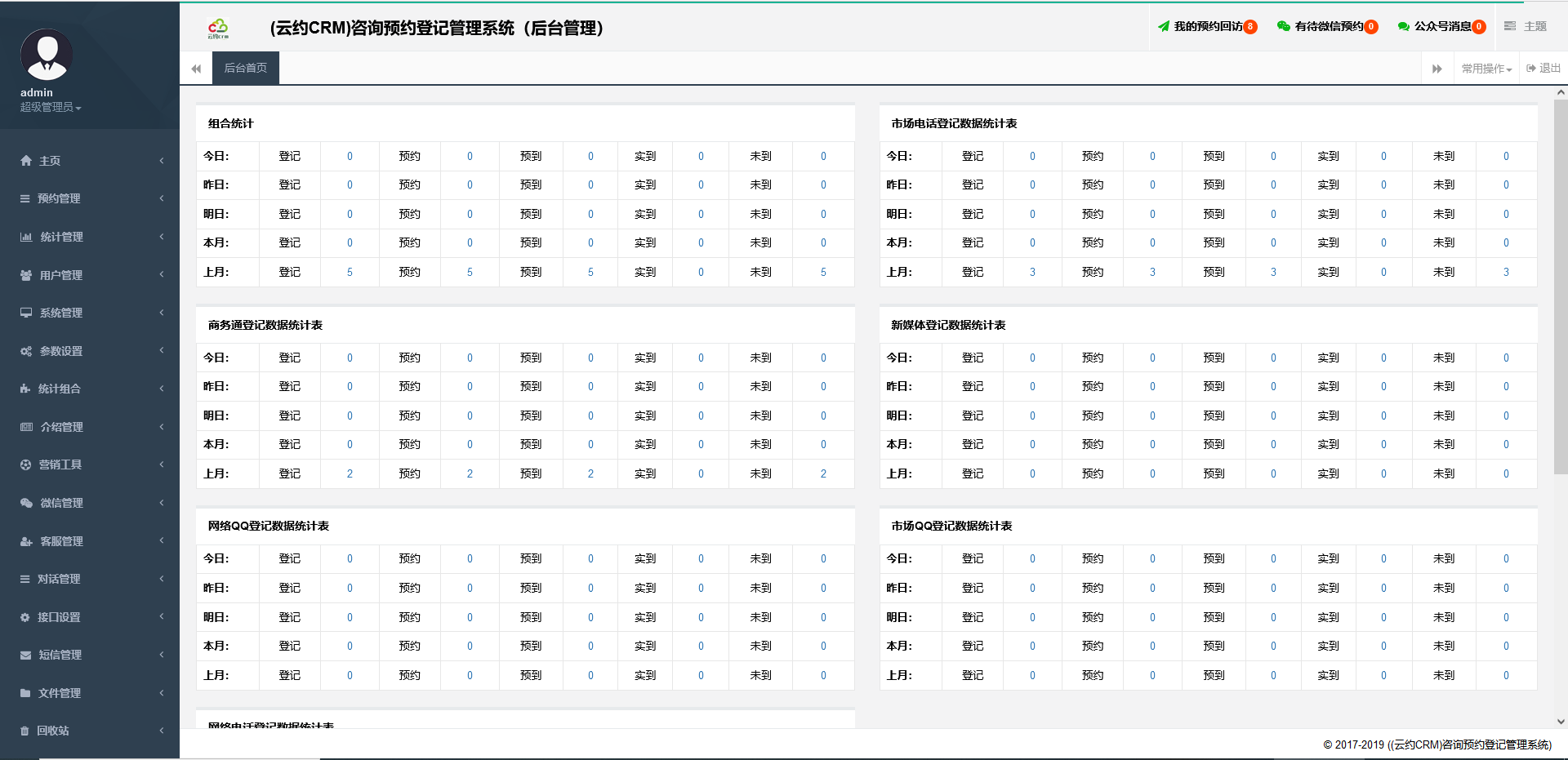 微信医疗(登记+咨询+回访)预约管理系统
微信医疗(登记+咨询+回访)预约管理系统  云约CRM微信小程序APP系统定制开发
云约CRM微信小程序APP系统定制开发 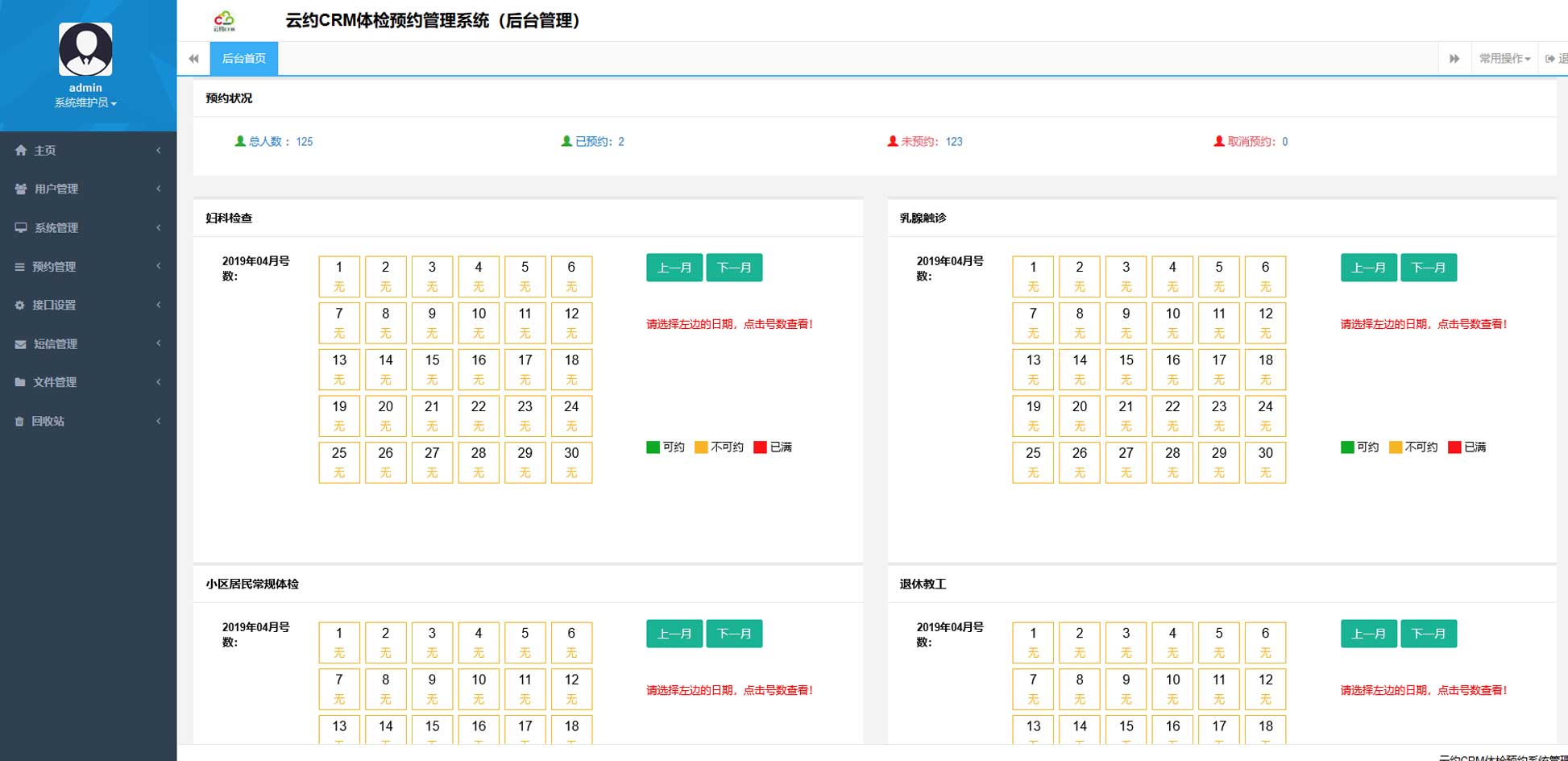 云约CRM体检自定义出号预约管理系统
云约CRM体检自定义出号预约管理系统  云约CRM云诊所系统,云门诊,医疗预约音视频在线问诊预约系统
云约CRM云诊所系统,云门诊,医疗预约音视频在线问诊预约系统 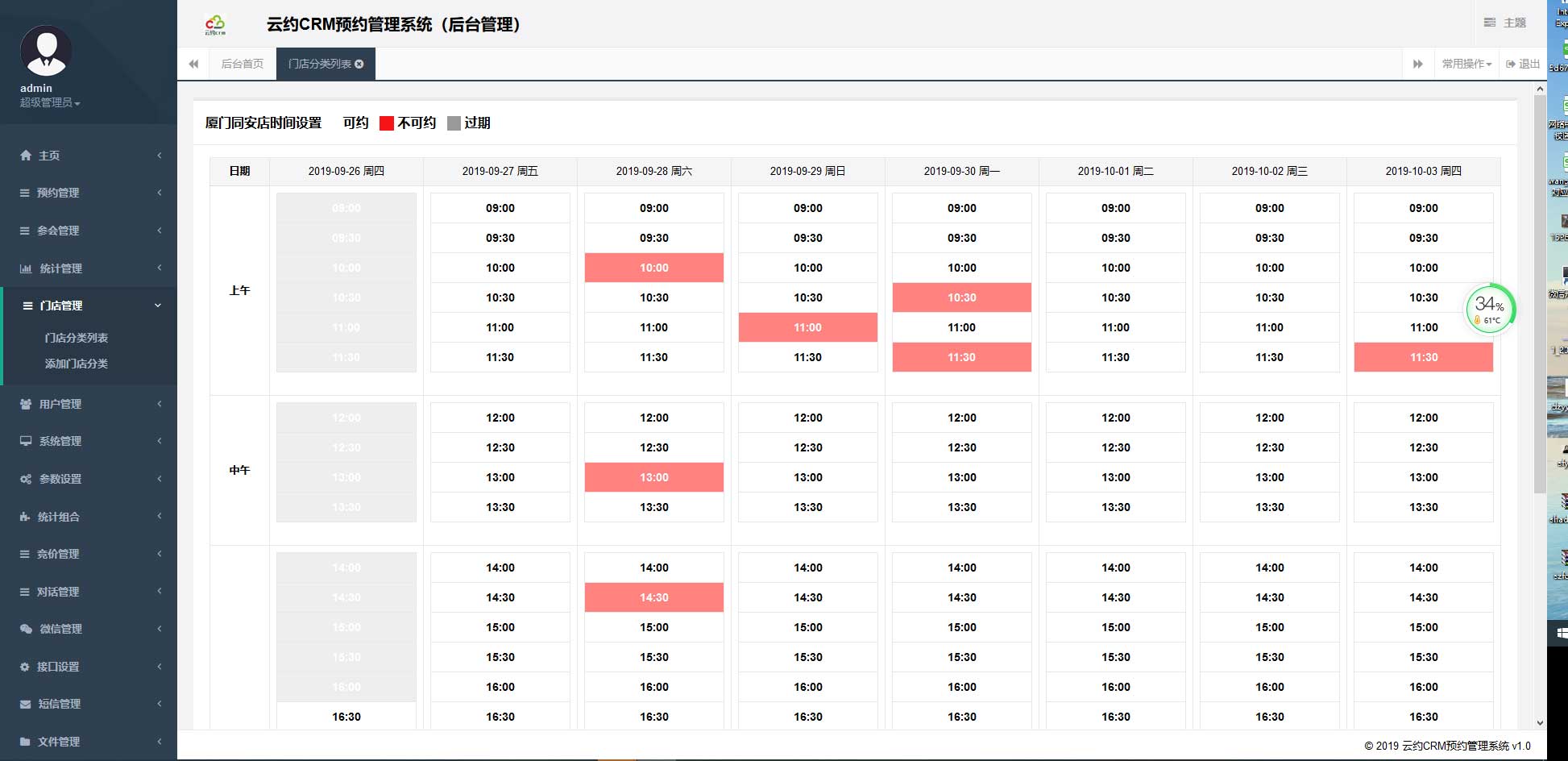 云约CRM新版美容微信预约系统门店版_门店预约管理系统
云约CRM新版美容微信预约系统门店版_门店预约管理系统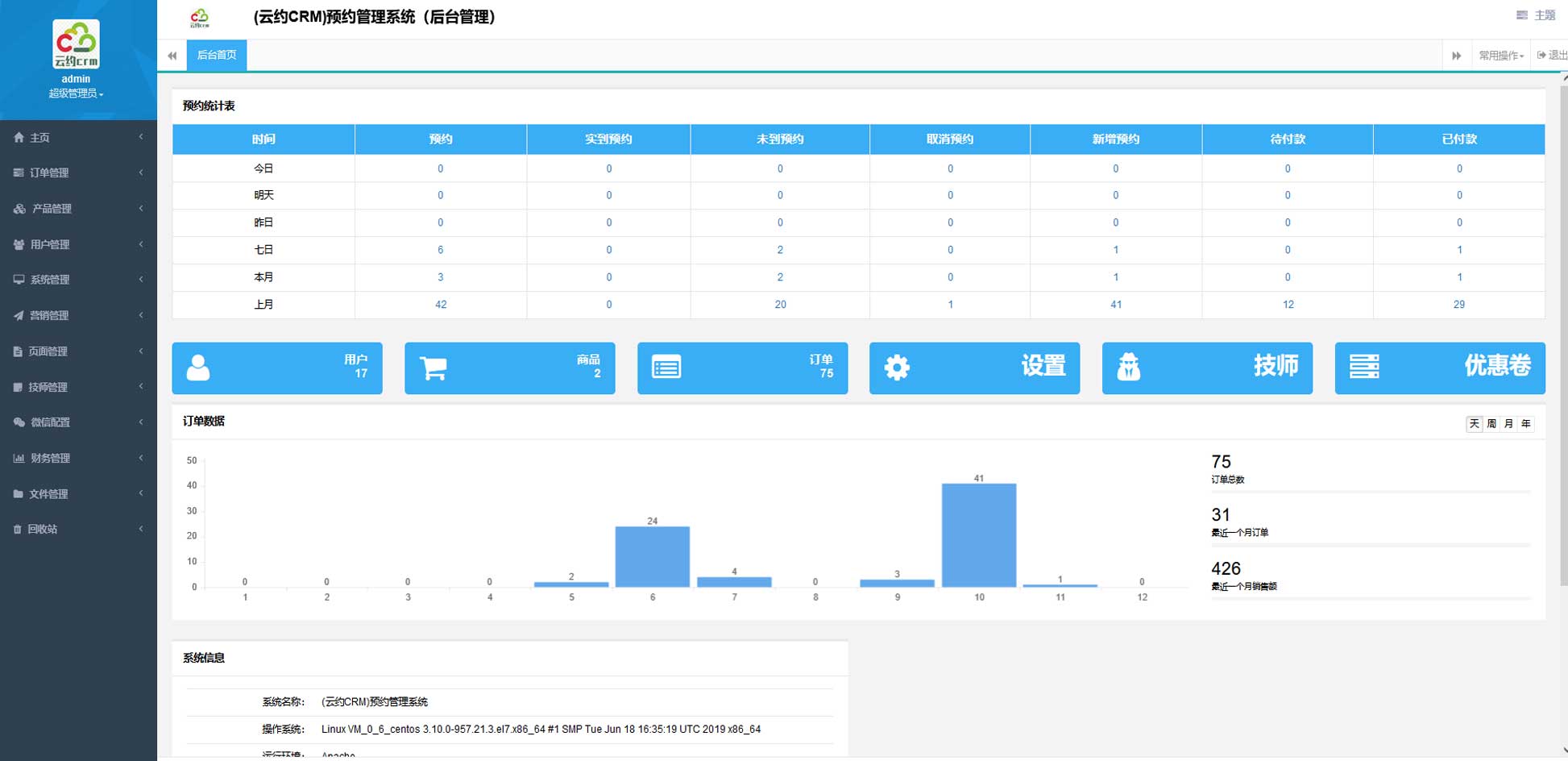 云约CRM最新ThinkPHP6通用行业的预约小程序(诊所挂号)系统
云约CRM最新ThinkPHP6通用行业的预约小程序(诊所挂号)系统联系电话:18300931024
在线QQ客服:616139763
官方微信:18300931024
官方邮箱: 616139763@qq.com