TargetScan特别擅长于预测人类及哺乳动物miRNA的靶点targetscan,通过识别与miRNA种子区域相匹配的保守碱基位点来实现主要功能 预测miRNA与靶基因的结合位点TargetScan能够预测miRNA与靶基因mRNA的潜在结合位点targetscan,这些位点通常位于mRNA的3#39非翻译区3#39UTR预测基于保守的碱基配对原则targetscan,特别是miRNA种子区域的78个。
targetscan是一个功能强大的在线工具,专门用于预测miRNA的靶基因在miRNA研究日益受到重视的今天,targetscan凭借其独特的算法特点和便捷的操作方式,成为targetscan了科研人员不可或缺的研究助手一targetscan的算法特点 targetscan将RNA间相互作用的热力学模型与序列比对分析相结合,这一创新性的方法使得其在预测miRNA。
在线预测miRNA靶基因的工具targetscan因其便捷性和准确性备受关注随着miRNA研究的持续升温,如2022年科学研究的活跃度,targetscan作为不可或缺的工具,只需通过网页操作即可实现miRNA靶基因的预测相较于其他软件,targetscan的独特之处在于它结合了RNA间相互作用的热力学模型与序列比对,引入信号信噪比评估预。
RNAhybrid输入文件格式与miRanda相同,需为FASTA格式运行预测工具 TargetScan下载并安装Perl脚本,使用命令行运行预测,如perl targetscan_70pl miR_9606_infotxt SEMA3Bfa target_ResultstxtmiRanda从GitHub下载并编译安装后,在src目录下运行miranda miRNA9606fa SEMA3Bfa grep #39。
操作流程登录官网用户需首先登录targetscan的官方网站targetscanorgvert_72选择物种在首页选择研究物种,目前targetscan支持多种生物种类的miRNA靶基因预测输入miRNA用户输入感兴趣的miRNA,并点击submit开始运行预测程序下载结果运行完毕后,用户可以下载预测结果,结果提供TXT和xlsx格式,方便用户。
首先,TargetScan基于miRNA和mRNA之间的结合模式进行预测它利用RNA二级结构分析,寻找miRNA与mRNA 3’UTR区域可能结合的区域其次,预测依据包括保守的种子区域匹配和互补性评估种子区域是miRNA的关键部分,与mRNA结合时起主要作用种子区域的匹配程度和互补性评估是预测靶基因的重要依据最后,TargetScan还。

TargetScan是一款专门用于预测miRNA结合位点的软件,对于哺乳动物中miRNA结合位点的预测效果极为出色为了进行miRNA靶基因预测,首先需要确定转录本的3’UTR区域,TargetScan数据库通过一种名为3Pseq的测序技术,来确定转录本对应的3’UTR区此技术结合分析结果和NCBI中已有的3’UTR注释,提供了一个综合的3。
1 访问TargetScan网站并输入查询信息首先,您需要访问TargetScan网站并输入您要研究的miRNA序列这个过程需要一些基本的计算机技能,包括了解文件类型和如何在网站上完成输入2 预测结果的输出一旦您完成了输入,TargetScan会输出一个结果页面,其中包含了对miRNA可能的靶向mRNA的信息这些结果可能包括。
TargetScan预测结果的解读主要关注以下几个方面种子区域匹配程度核心点种子区域是miRNA与mRNA结合的关键部位,匹配程度直接影响调控效果解读TargetScan会给出种子区域与miRNA的匹配得分,得分越高表示匹配程度越好,miRNA对该目标mRNA的调控可能性越大结合位点的保守性核心点保守性高的结合位点意味。
链接 图片PITA 简介PITA数据库基于靶位点的可接性和自由能来预测miRNA的靶标特点主要包含人小鼠果蝇和蠕虫的信息,允许对靶关系的预测和下载链接mir07_dyn_datahtml 图片miRTarBase 简介miRTarBase主要收集。
TargetScan结果表格一般包括以下列内容及其解释基因名称 说明显示目标基因的名称,是识别和分析特定基因的重要标识靶点序列 说明表示与特定基因相关的调控序列或miRNA靶向序列,这些序列是分析的关键,因为它们代表了潜在的调控点,可能影响基因的表达评分 说明反映预测的可靠性或结合强度的数值,基于。
常用的microRNA靶基因预测工具包括以下几种TargetScan 网址新版 targetscanorgvert_72 或老版 targetscanorgmamm_31特点操作简单,可以输入基因名查询其3’UTR可能作用的多个miRNA,或输入miRNA名查询可能作用的多种靶基因最新版增加了多种3’UTR的展示miRcode 网址mircodeorgindex。
而kmer则是指将核酸序列分成包含k个碱基的字符串,即从一段连续的核酸序列中迭代地选取长度为K个碱基的序列,若核酸序列长度为L,kmer长度为K,那么可以得到LK+1个kmers如下图所示,假设这里存在某序列长度为21,设定选取的kmer长度为7,则得到217+1=15个7mers 而targetscan 中。
Targetscan结果表格的解读相对直观,主要通过分析基因名称来初步评估其功能和可能的生物学意义首先,你需要在表格中定位到你感兴趣的特定基因,这一步可能依赖于你的研究目标或者已知的相关基因找到后,仔细查看该条目的详细信息,包括其对应的靶点预测调控模式等接着,关注quot文献报道quot部分,这将为你。

一生物信息学方法 生物信息学方法主要依赖于计算机算法来预测miRNA的靶基因这些算法通过分析miRNA序列与mRNA 3#39UTR序列的互补性保守性热稳定性以及靶位点处的二级结构等因素,来预测潜在的靶基因常用算法TargetScan通过搜索与每个miRNA的种子区匹配的保守位点来预测靶基因,并提供每个miRNA预测。
有很多生物软件可以预测某个miRNA的靶基因,如TargetScan,mirBase,PicTar,结合miRNA芯片筛选确定靶蛋白后,通过overexpression或antisense改变该miRNA的表达量,QRTPCR及Western Blot检测相应蛋白含量的变化将预测靶蛋白的启动子转载到pGL3载体,转染细胞并检测luciferase的变化倍数最后通过IP证明两者间的结合。
预测miRNA的靶基因主要可以通过以下几种方法一基于序列匹配的方法 这类方法的核心在于比对miRNA序列与靶基因的3#39非翻译区3#39 UTR序列,寻找具有互补配对的miRNA靶位点常用的工具包括TargetScan一个广泛使用的miRNA靶基因预测工具,它基于序列匹配和保守性来预测miRNA的靶基因miRanda另一个。
下一篇: ie缓存清理,ie缓存怎么清理 彻底
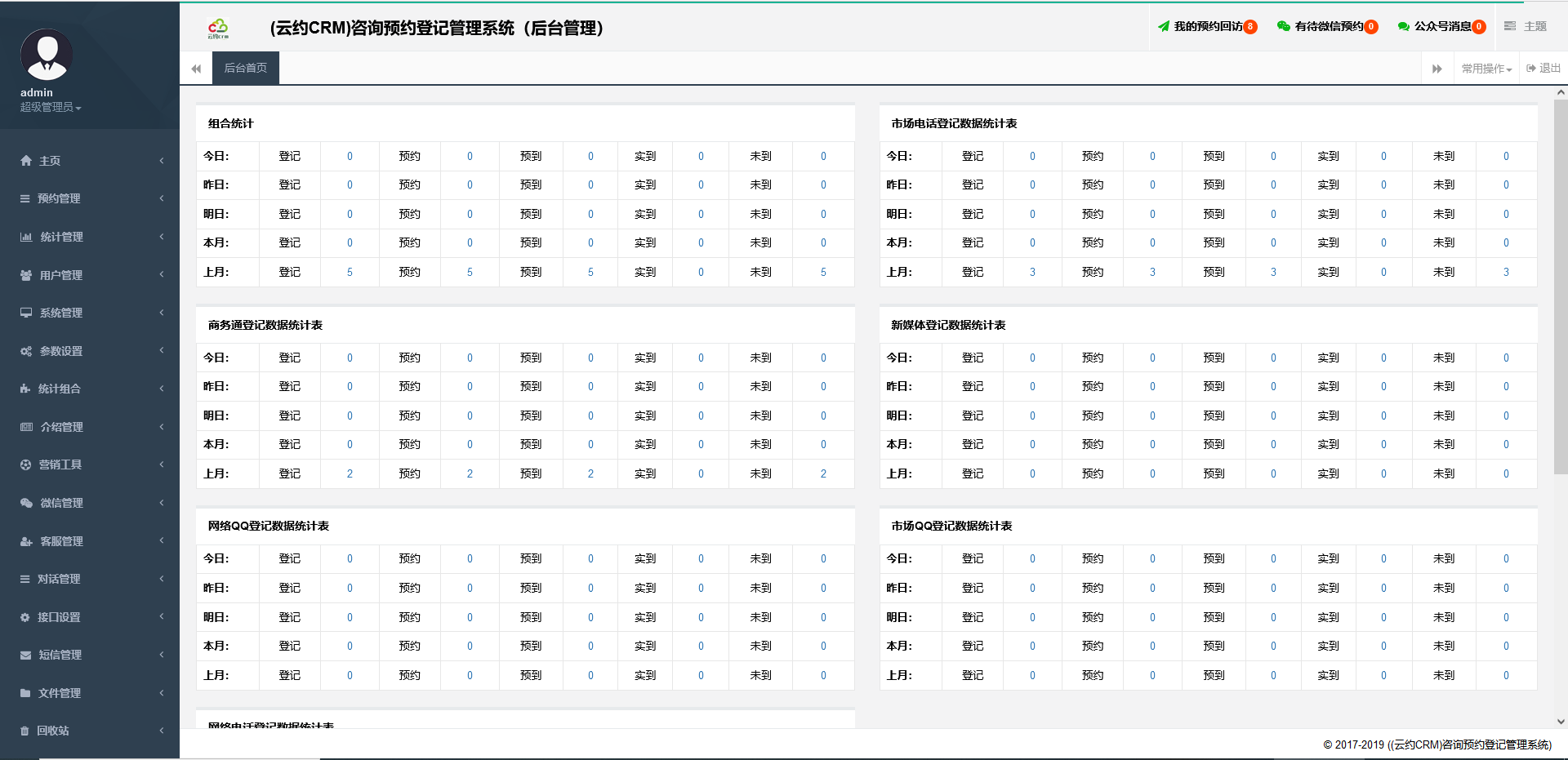 微信医疗(登记+咨询+回访)预约管理系统
微信医疗(登记+咨询+回访)预约管理系统  云约CRM微信小程序APP系统定制开发
云约CRM微信小程序APP系统定制开发 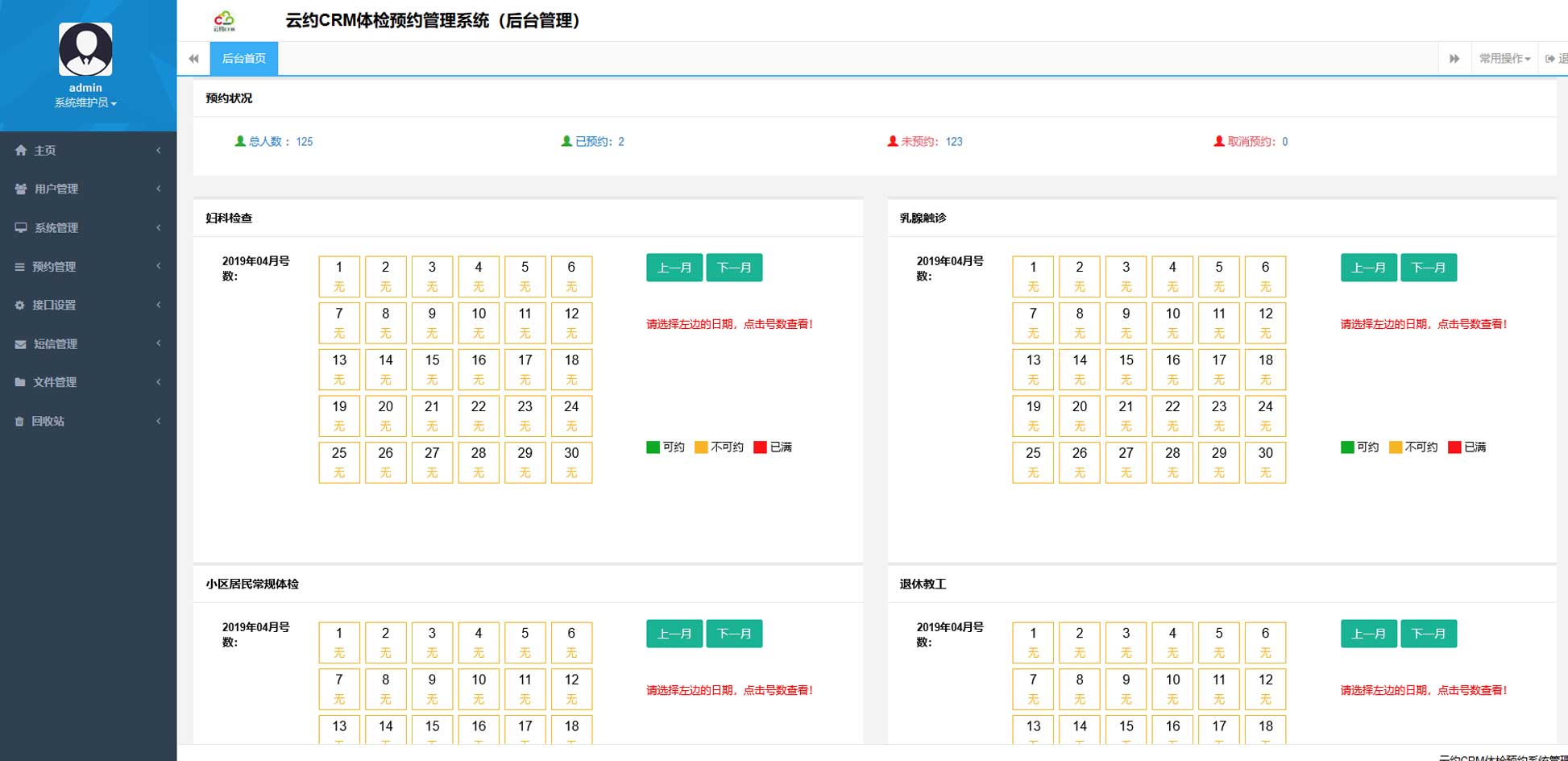 云约CRM体检自定义出号预约管理系统
云约CRM体检自定义出号预约管理系统  云约CRM云诊所系统,云门诊,医疗预约音视频在线问诊预约系统
云约CRM云诊所系统,云门诊,医疗预约音视频在线问诊预约系统 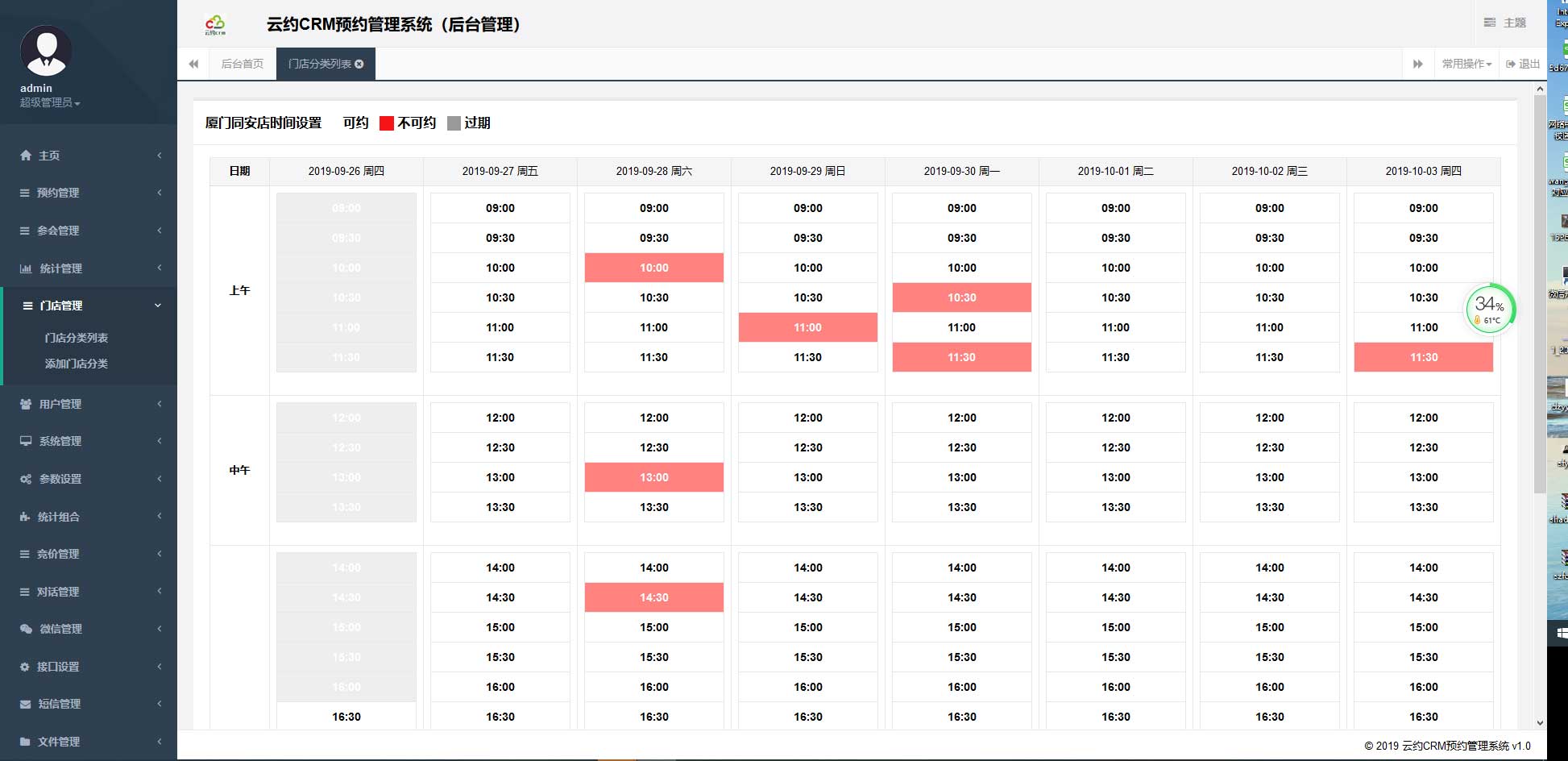 云约CRM新版美容微信预约系统门店版_门店预约管理系统
云约CRM新版美容微信预约系统门店版_门店预约管理系统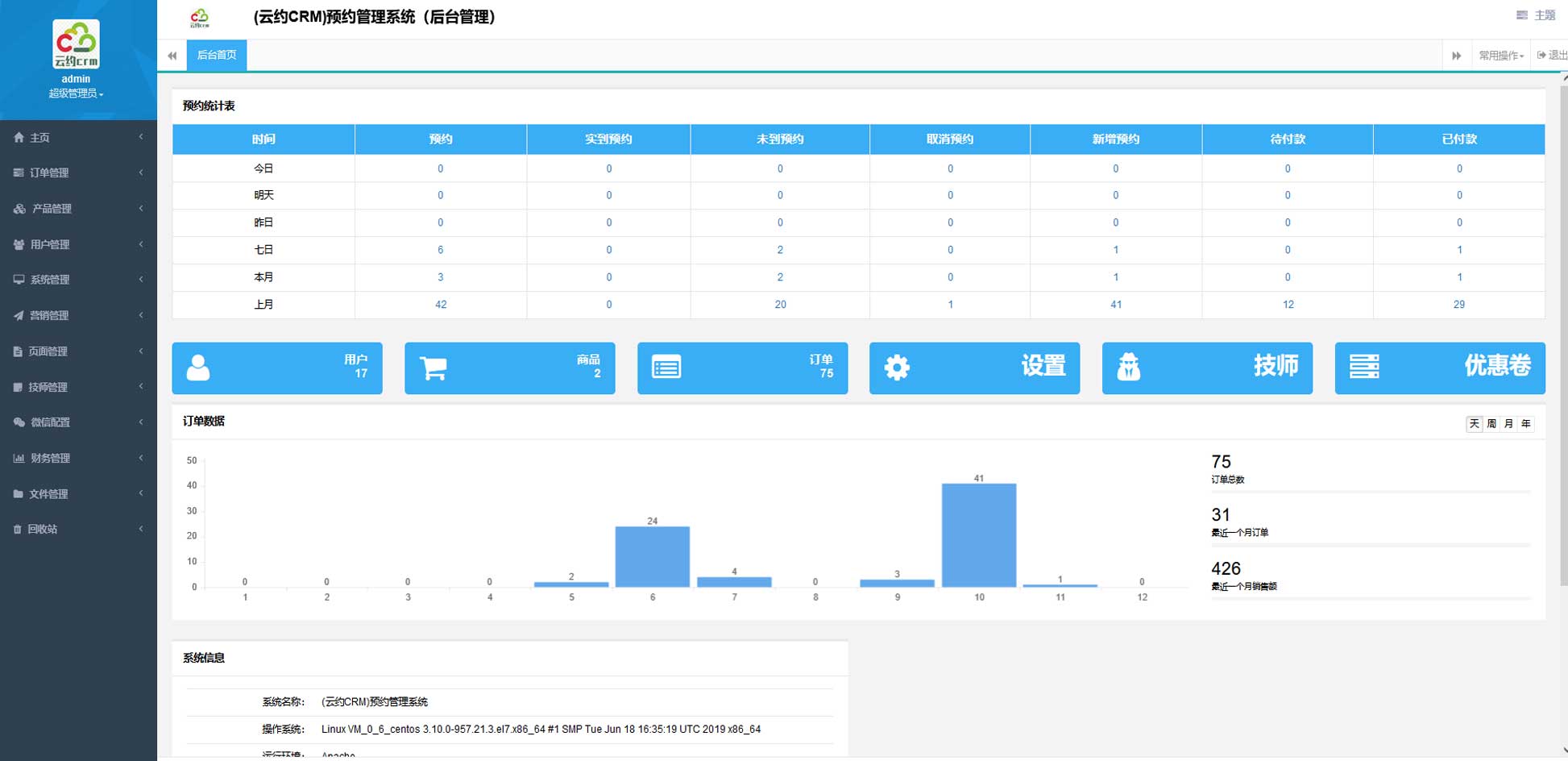 云约CRM最新ThinkPHP6通用行业的预约小程序(诊所挂号)系统
云约CRM最新ThinkPHP6通用行业的预约小程序(诊所挂号)系统联系电话:18300931024
在线QQ客服:616139763
官方微信:18300931024
官方邮箱: 616139763@qq.com