结合位点预测工具如TargetscanStarbasemirna靶基因分析,预测结合位点合成miRNA mimics,扩增目mirna靶基因分析的片段,构建载体,设置转染组别,验证miRNA与靶基因互作去除背景值,数据归一化,分析得出结果重庆医科大学廖正步教授研究发现损伤脑组织中circIgfbp2显著升高,敲低可减轻TBI小鼠的焦虑和睡眠障碍研究发现circIgfbp2与hsamiRmirna靶基因分析;预测miRNA靶基因的软件如targetscan和ENCORI,分别通过不同算法,如RNA相互作用分析,实现对miRNA与mRNAlncRNAcircRNA等调控关系的预测利用R语言编写代码,结合数据库数据,实现批量预测,便于深入分析调控网络为了使学习者系统掌握相关知识,作者整理了一门线上课程课程内容涵盖了miRBasemiRTarBase。
通过对比实验组与对照组的荧光素酶活性变化,分析 miRNA 对靶基因表达的调控作用数据分析与结果解读 在数据分析阶段,首先去除空白对照中的背景值,将萤火虫荧光素酶数值减去空白对照值,海肾荧光素酶数值也做相同处理接着,将去除背景后的萤火虫荧光素酶数值除以去除背景后的海肾荧光素酶数值,得到归一;5 miRDB 在线数据库,用于miRNA目标预测和功能注释,适用于人类小鼠等五个物种6 Starbase 分析32种癌症类型的相关数据,提供泛癌网络,适用于不同组学的数据挖掘7 miRTarBase 收集超过36万条miRNA靶标相互作用,提供实验验证数据集8 TarBase 收录人和小鼠的实验验证miRNA靶基因信息,提供高。
2实时定量分析表明木薯中的7个miRNA受低温诱导表达,且它们在C4,和SC124品种中及不同的低温处理方式下都具有差异表达其中2个miRNAmiRNA5和miRNA2的靶基因在两品种和不同低温处理方式下也具有差异表达3通过对2个miRNA和它们靶基因的定量表达分析发现,无论在C4和SC124品种中还是;合成microRNA mimics和 microRNA negativecontrol准备细胞转染,将构建的载体与microRNA相关产品共转染细胞荧光素酶检测,共转染细胞2472小时后,通过检测确定目的microRNA的靶基因解决实验中常见问题 1 如何判断转染成功通过luc读值判断3#39 UTR质粒及Renilla质粒转染成功对于miRNA质粒或mimic,可。
因此,结合实验验证是确保miRNA靶基因鉴定准确性不可或缺的环节总之,寻找miRNA的靶基因是一个综合了理论与实践的过程通过精确的生物信息学分析和严谨的实验验证,科学家们正逐步揭开miRNA调控基因表达的复杂面纱,为疾病治疗和基因调控机制的研究开辟新的道路。
TargetScan是一款广受认可的miRNA靶基因预测工具,其预测原理基于多个关键点首先,TargetScan基于miRNA和mRNA之间的结合模式进行预测它利用RNA二级结构分析,寻找miRNA与mRNA 3’UTR区域可能结合的区域其次,预测依据包括保守的种子区域匹配和互补性评估种子区域是miRNA的关键部分,与mRNA结合时起主要作用。
研究miRNA基因通常包括获取miRNA和其功能研究两大部分获取miRNA具体步骤为通过总RNA分离20 nt左右小分子RNA,接头增加其长度进行逆转录,PCR扩增,胶回收,克隆测序分析得到microRNAsmiRNA功能研究常用方法有超表达microRNA的前体和开发短串联模拟靶标技术STTMSTTM技术能阻碍microRNA活性,抑制其与靶。
基于序列的预测工具主要包括以下几种SequenceBased Target Prediction Tools这些工具的共同特点是,它们不需要miRNA的ID信息,而是通过序列比对和分析,预测miRNA与目标基因之间的相互作用这类工具的广泛应用,使得研究人员能够在没有充分ID信息的情况下,进行高效的miRNA靶基因预测为了解决公众号内容分散。
symbol和EntrezID,但需注意单次输入基因ID或symbol数量限制,以确保数据准确miRWalk20不仅简化了miRNAmRNA相互作用研究的流程,还提供了多维度的数据分析,助力科研人员深入探索miRNA生物学功能通过合理利用该数据库提供的资源,研究者可以更高效地预测和验证miRNA靶基因,推动相关领域研究的进展。

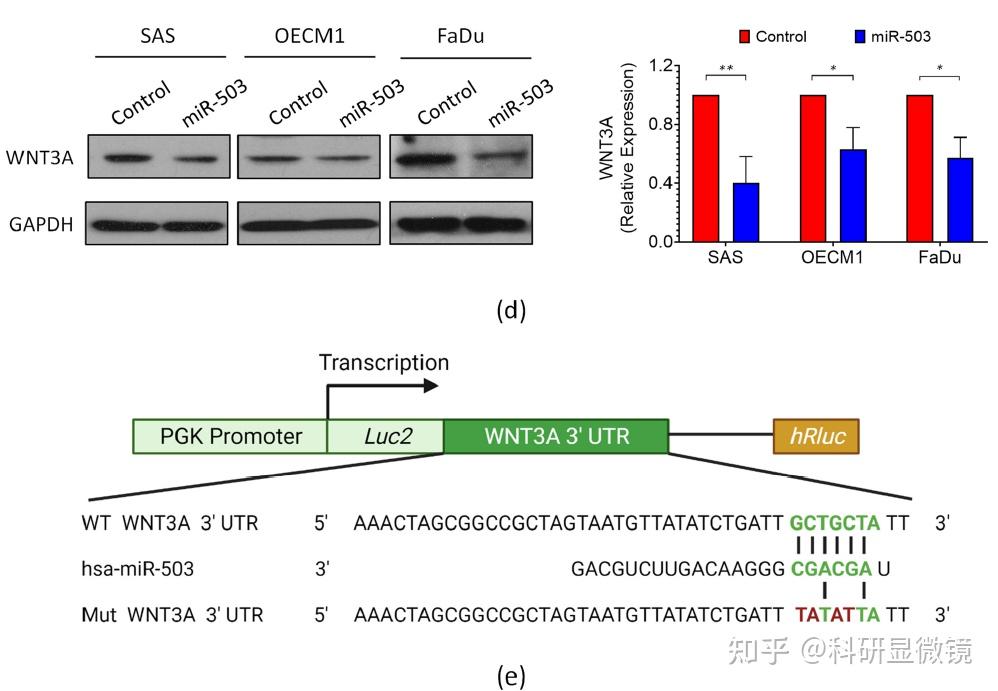
miRNA与靶基因的互作实验验证方法主要包括以下几种双荧光素酶报告基因系统该方法通过构建含有miRNA靶标位点的Luciferase报告基因载体,并将其转染至细胞中,然后共转染miRNA和Luciferase报告基因载体miRNA结合到靶基因的3#39UTR区域,可能会导致Luciferase基因表达抑制,进而通过测量Luciferase活性来确定miRNA和靶。
首先,基于序列匹配的方法通过比较miRNA序列与靶基因3#39非翻译区3#39 UTR序列,寻找互补配对的miRNA靶位点常用工具如TargetScanmiRandaPicTar等其次,基于保守性的方法利用跨物种基因组序列比对,预测miRNA靶位点上的保守序列区域,识别可能的靶基因PhyloPPhastCons等工具适合进行此类保守性分析再。
首先,采用双荧光素酶报告基因实验来判断miRNA是否与靶基因结合这一步通常通过将预测的能够与miRNA结合的靶基因3’UTR序列插入载体,并与萤火虫荧光蛋白的3’UTR结合当miRNA与插入序列结合时,会抑制萤火虫荧光蛋白的翻译,导致荧光值的降低,以此评估两者之间的结合情况其次,pulldown实验是另一种。
实验方法也是寻找miRNA靶基因的重要途径研究人员通过基因表达分析芯片或测序技术,提高或抑制miRNA活性,观察mRNA表达变化,从而寻找靶基因此外,蛋白质组学方法,如SILAC或2DDIGE技术,通过检测转染miRNA后蛋白表达水平的变化,提供靶基因信息免疫沉淀技术,尤其是紫外交联免疫沉淀结合高通量测序HITS。
目前最常用的方法是荧光素酶报告基因法首先,构建荧光素酶表达载体,将希望鉴定的miRNA靶基因的3’UTR插入荧光素酶基因的3’UTR中,然后将构建好的重组载体转染到细胞中,并改变miRNA的表达水平,最后检测荧光素酶的表达情况,以分析3’UTR中是否含有miRNA的靶位点目前有多家公司提供荧光素酶报告载体。
 微信医疗(登记+咨询+回访)预约管理系统
微信医疗(登记+咨询+回访)预约管理系统  云约CRM微信小程序APP系统定制开发
云约CRM微信小程序APP系统定制开发  云约CRM体检自定义出号预约管理系统
云约CRM体检自定义出号预约管理系统  云约CRM云诊所系统,云门诊,医疗预约音视频在线问诊预约系统
云约CRM云诊所系统,云门诊,医疗预约音视频在线问诊预约系统 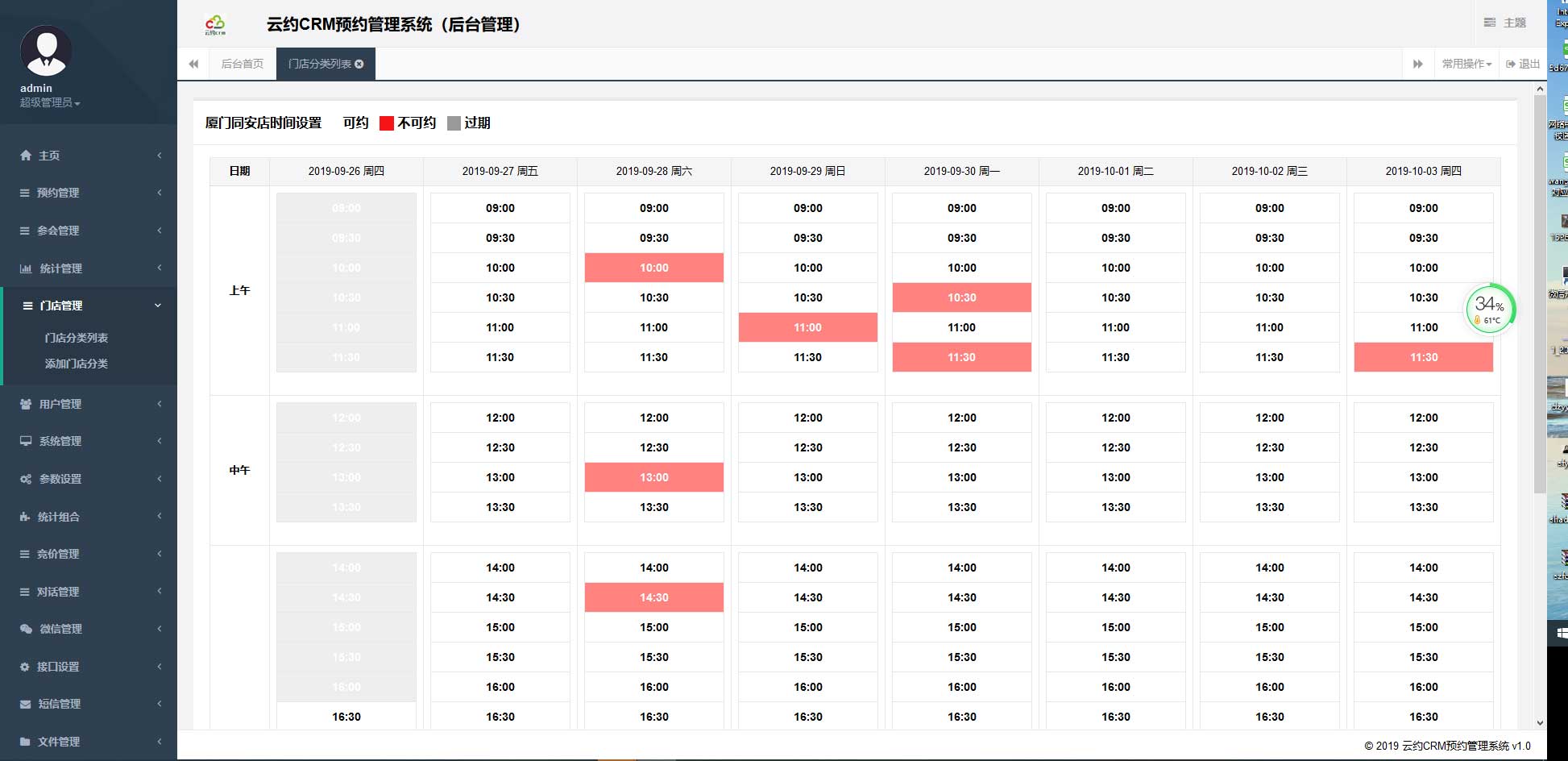 云约CRM新版美容微信预约系统门店版_门店预约管理系统
云约CRM新版美容微信预约系统门店版_门店预约管理系统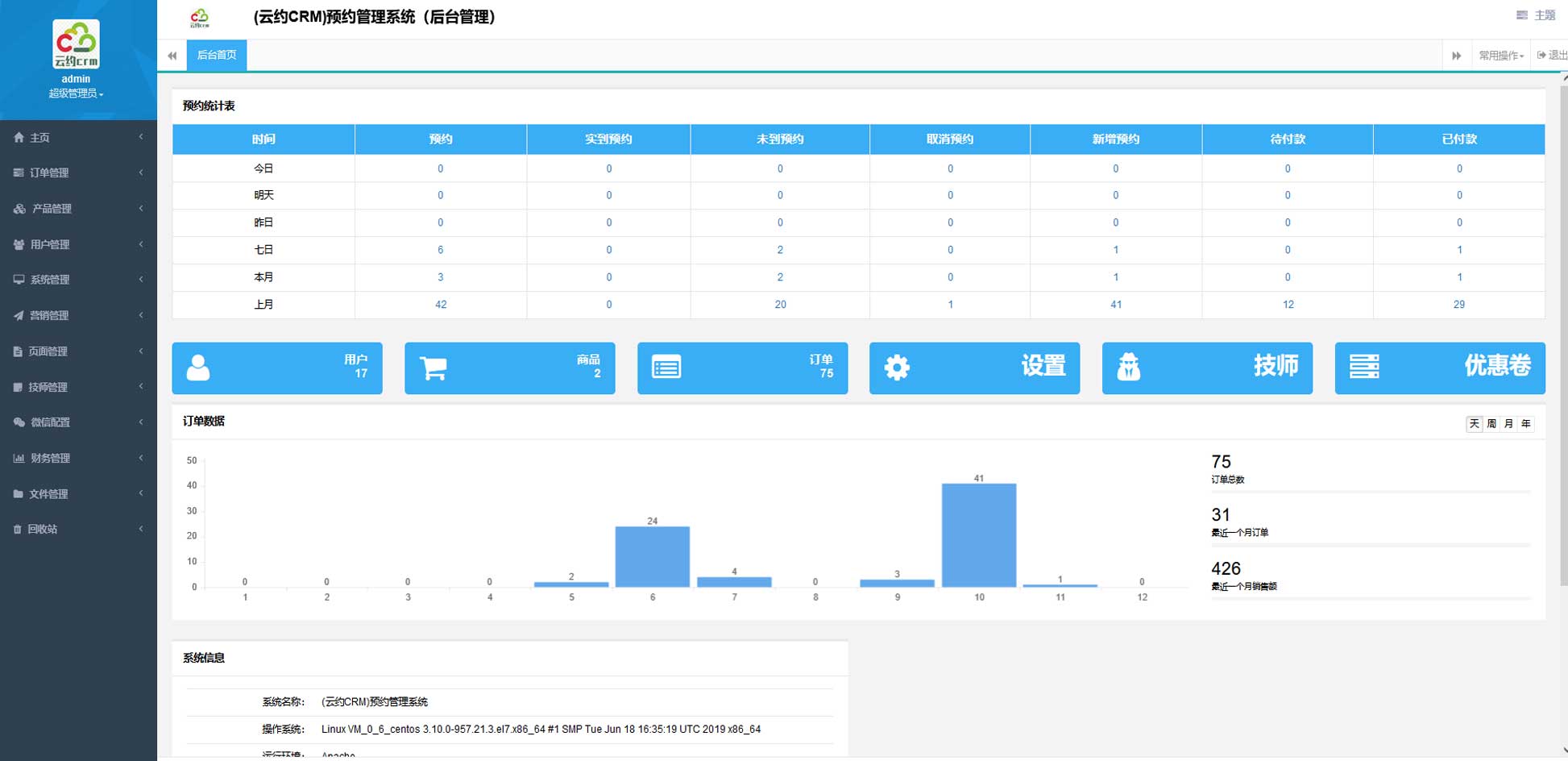 云约CRM最新ThinkPHP6通用行业的预约小程序(诊所挂号)系统
云约CRM最新ThinkPHP6通用行业的预约小程序(诊所挂号)系统联系电话:18300931024
在线QQ客服:616139763
官方微信:18300931024
官方邮箱: 616139763@qq.com